Zweigerdt Lab
Unter Verwendung humaner pluripotenter Stammzellen (hPSCs) arbeiten wir an Aspekten der Entwicklungsbiologie und der regenerativen Medizin mit Schwerpunkt auf dem Herzen sowie anderen Geweben und Zelltypen wie Makrophagen. Die Forschung ist in verschiedene, aber eng miteinander verknüpfte Themen und Projekte gegliedert:
1. Entwicklung von Bioprozessen für die klinische Umsetzung von hPSC-abgeleiteten Zelltypen, insbesondere von Mesendoderm-Derivaten wie Kardiomyozyten, Endothelzellen, Makrophagen und definitivem Endoderm.
2. Mechanismen der Mesendoderm-Spezifikation und der Herzentwicklung, wie z. B. die In-vitro-Induktion, Charakterisierung und Stabilisierung kardialer Vorläuferzellen (SCPs) und die Rekapitulation früher Schritte der nativen Herzentwicklung in vitro mittels Herzbildender Organoide (HFOs), die das funktionelle Zusammenspiel zwischen Herzanlage und Vorderdarm-Endoderm nachbilden.
3. Modellierung von (angeborenen) Herzerkrankungen in der Schale. Unsere Projekte in diesem Bereich umfassen elektrophysiologische Störungen (z. B. Brugada-Syndrom aufgrund von Mutationen im SCN5A-Gen), die Störung kardialer Transkriptionsfaktoren (z. B. NKX2.5 Knock Out in HFOs) und die Untersuchung der kardialen Hypertrophie in hPSC-Kardiomyozyten auf der Grundlage pharmakologischer Stimuli oder genetischer Mutationen (z. B. in MYH7 oder MYBPC3).
Zusammenarbeit mit der Industrie: Neben akademischen Partnern (innerhalb und außerhalb der MHH; unten in Bezug auf spezifische Projekte aufgeführt) arbeiten wir bei vielen unserer Forschungsprojekte mit Industriepartnern zusammen, darunter DASGIP-Eppendorf, LaVision BioTec, Miltenyi Biotec, Novo Nordisk, Thermo Fisher und Thinking Research Instruments (in alphabetischer Reihenfolge).
1. Bioprozessentwicklung für die klinische Umsetzung von hPSC- abgeleiteten Zelltypen
In den letzten Jahren haben mehrere hPSC-basierte Therapiekonzepte Fortschritte in Richtung erster Studien am Menschen gemacht. Dieser Fortschritt geht einher mit dem Bedarf an effizienten und robusten Kultur- und Differenzierungsstrategien zur Herstellung von hPSCs in klinisch relevanter Menge, Qualität und zu kommerziell vertretbaren Bedingungen.
Unsere Vision für die klinische und industriekonforme Produktion von hPSC-abgeleiteten Zelltypen konzentriert sich auf den Einsatz von Rührkessel-Bioreaktortechnologien (STBR), um Robustheit, Skalierbarkeit, Automatisierung und letztlich GMP-konforme Zellproduktionsprozesse zu gewährleisten. Dieses Konzept umfasst die Kultur, Massenproduktion und Differenzierung von hPSCs als völlig matrixfreie, reine Zellaggregate in gerührter Suspensionskultur. Die Strategie ermöglicht eine rationale Prozessoptimierung durch In-silico-Modellierung, Upscaling durch einen Seed-Train-Ansatz und eine Closed-Loop-Produktion, die für GMP-Konformität und Automatisierung erforderlich ist. (Abbildung 1).
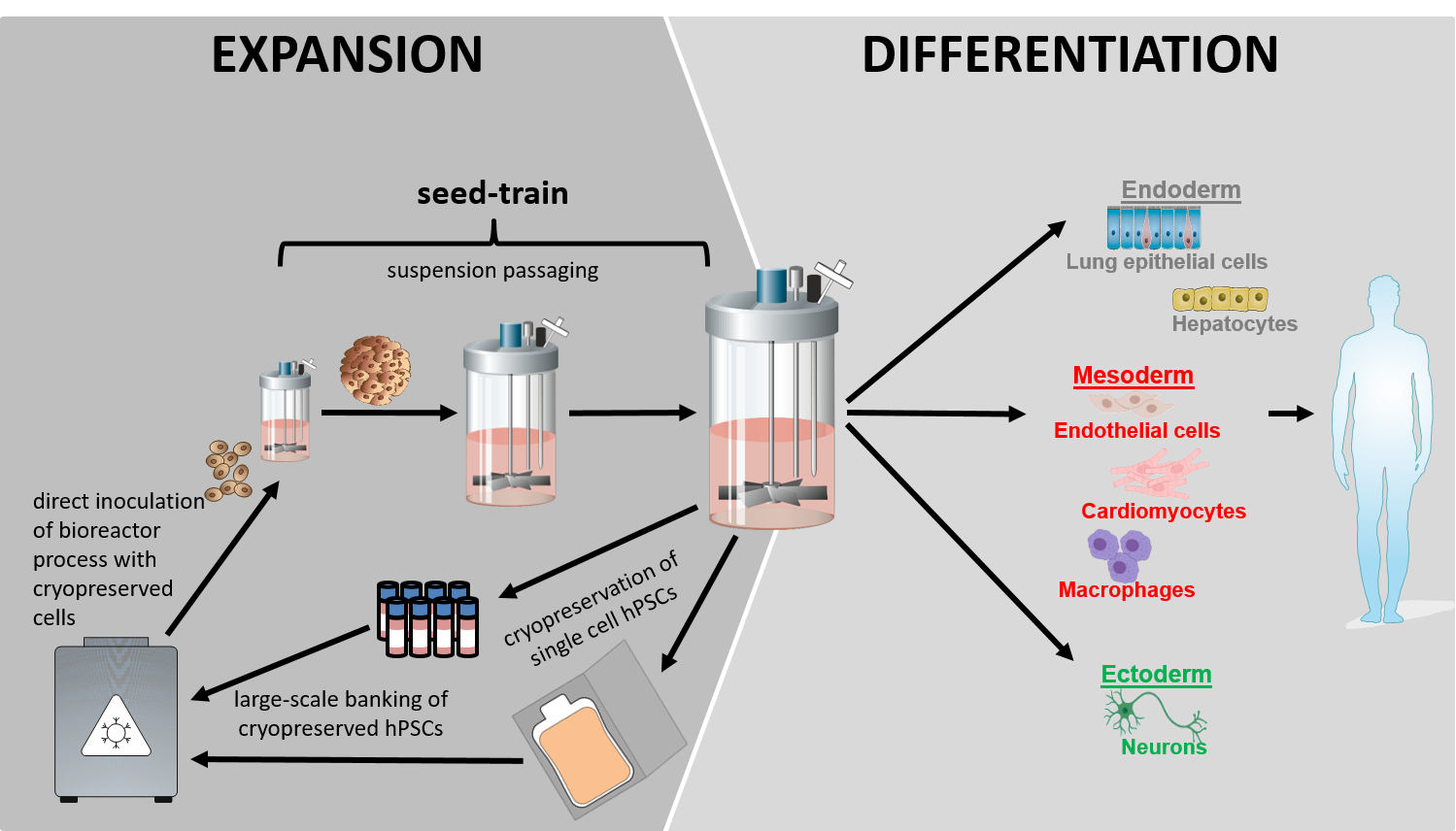
Aktuelle Entwicklungen:
- Herstellung von kryokonservierten Zellbanken für die direkte Suspensionskultur in STBRs, wodurch eine vollständig matrixfreie hPSC-Kultur ermöglicht wird.
- Förderung der Prozesseffizienz durch In-silico-Prozessmodellierung sowohl für die hPSC-Expansion als auch für die linienspezifische Differenzierung.
- Verbesserung des Ansatzes der kardiomyogenen Differenzierung und der Prozesskontrolle durch Sekretomanalyse.
- Ermöglichung von Closed-Loop-Bioprozessen für die GMP-konforme Zellproduktion.
Kooperationen und Projekte zur Bioprozessentwicklung:
Einerseits konzentrieren wir uns auf das Upscaling und die Optimierung von Prozessen für die Herstellung pluripotenter Zell-Aggregate, die als "Rohmaterial" für die liniengerichtete Differenzierung(en) dienen.
Unser zweiter Schwerpunkt liegt auf der gerichteten kardiomyogenen Differenzierung von hPSCs. In Zusammenarbeit mit den MHH-Gruppen von Ulrich Martin (LEBAO), Axel Haverich (HTTG) und zahlreichen anderen Forschern werden die aus dem Prozess gewonnenen Kardiomyozyten für die klinische Herzreparatur im iCARE-Projekt (LINK) evaluiert. Die Anwendung von hPSC-abgeleiteten Kardiomyozyten und Endothelzellen - letztere werden von der Gruppe von Ruth Olmer (MHH/ LEBAO) generiert - erfolgt in Zusammenarbeit mit den LEBAO-Gruppen von Ina Gruh, Andres Hilfiker/Birgit Andree, Andreas Martens und Thomas Aper im Projekt 3D-Heart-2B, das auf das In-vitro-Engineering von mehrschichtigen, vaskularisierten, linearen Herzschlauchgewebekonstrukten abzielt (LINK).
Eine weitere wichtige Zusammenarbeit zielt auf die kontinuierliche Produktion von hiPSC-Makrophagen im großen Maßstab in enger Zusammenarbeit mit der Gruppe von Nico Lachmann (MHH) ab, die auch vom Industriepartner Novo Nordisk (Dänemark; www.mhh.de/en/presse/mhh-insight/news-detailed-view/cells-of-the-future) finanziert wird.
- Diana Massai (Politecnico di Torino, Italy)
- Elizabeth Lipke and Selen Cremaschi (Auburn University, US)
- DASGIP-Eppendorf (Hamburg, Germany)
- Thermo Fisher Scientific (Darmstadt, Germany)
- Thinking Research Instruments (Hamburg, Germany)
- Ackermann et al. Nat Protoc 2022 (https://www.nature.com/articles/s41596-021-00654-7 ),
- Manstein et al. STAR Protoc. 2021 (https://star-protocols.cell.com/protocols/1227 ),
- Manstein et al. Stem Cells Transl Med. 2021 (https://stemcellsjournals.onlinelibrary.wiley.com/doi/full/10.1002/sctm.20-0453 ),
- Williams et al. Front Bioeng Biotechnol 2020 (https://www.frontiersin.org/articles/10.3389/fbioe.2020.00851/full ),
- Halloin et al. Stem Cell Reports 2019 (https://www.sciencedirect.com/science/article/pii/S2213671119303303?via%3Dihub ),
- Sahabian et al. Cells 2019 (https://www.mdpi.com/2073-4409/8/12/1571 ),
- Ackermann et al. Nat Commun. 2018 (https://www.nature.com/articles/s41467-018-07570-7 ),
- Olmer et al. Stem Cell Reports 2018 (https://www.sciencedirect.com/science/article/pii/S2213671118301449?via%3Dihub ),
- Massai et al Sci Rep. 2017 (https://www.nature.com/articles/s41598-017-04158-x ),
- Kropp et al. Stem Cells Transl Med. 2016 (https://stemcellsjournals.onlinelibrary.wiley.com/doi/full/10.5966/sctm.2015-0253 ),
- Kempf et al. Nat Protoc. 2015 (https://www.nature.com/articles/nprot.2015.089 ),
- Kempf et al. Stem Cell Reports. 2014 (https://www.sciencedirect.com/science/article/pii/S2213671114003026?via%3Dihub ),
- Zweigerdt et al. Nat Protoc. 2011 (https://www.nature.com/articles/nprot.2011.318 ).
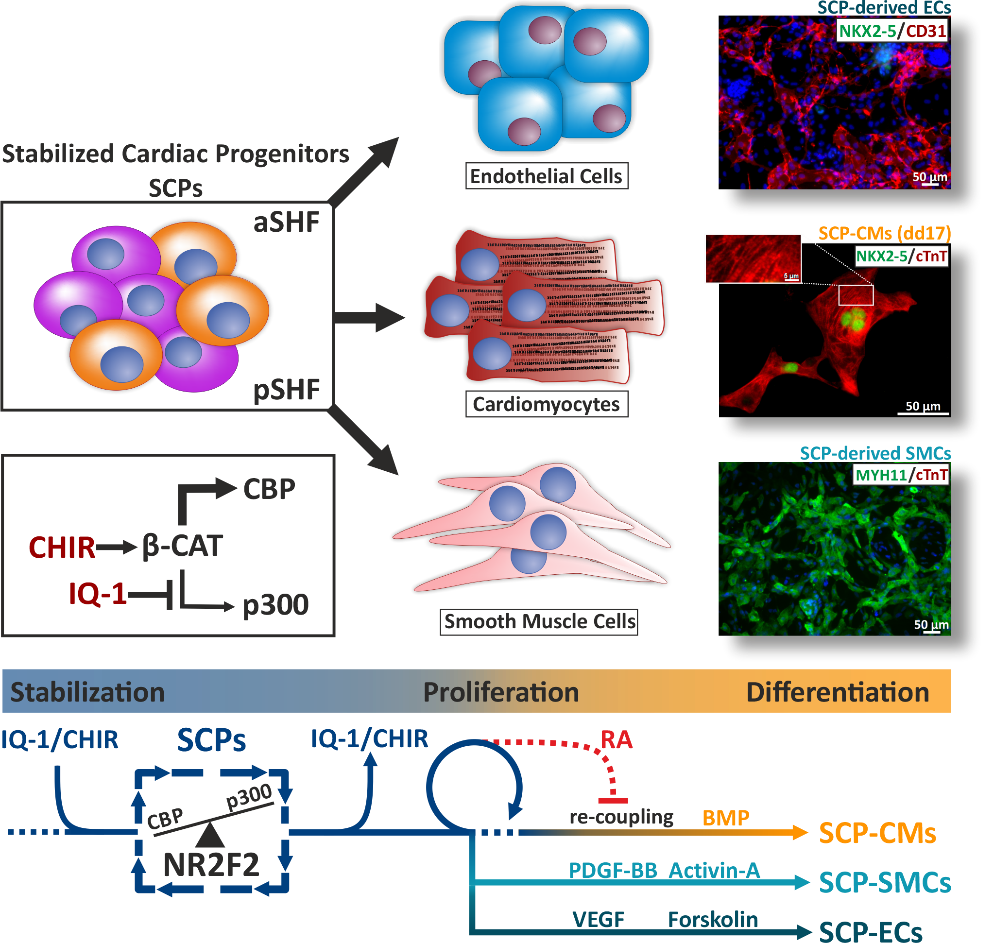
2. Mechanismen der Mesendoderm-Spezifikation sowie der Herzentwicklung und -regeneration
Ähnlich wie bei der Embryogenese ist der In-vitro-Übergang von der Pluripotenz der hPSCs zur Spezifizierung und Differenzierung der Zellen sehr komplex und kaum verstanden. Jüngste Arbeiten haben jedoch gezeigt, dass viele der komplexen molekularen und zellulären Mechanismen, die die native Entwicklung von Zellen, Geweben und Organen im Embryo steuern (die häufig in Modellorganismen wie Fröschen, Fischen und Mäusen untersucht werden), durch fortgeschrittene Strategien der hPSC-Differenzierung in vitro, einschließlich sogenannter "Organoide", genau rekapituliert und untersucht werden können. Wir konzentrieren uns auf die Spezifizierung des Mesendoderms und die Herzentwicklung und arbeiten an mehreren Projekten in diesem spannenden Bereich der hPSC-Forschung.
Kardiale Vorläuferzellen sind während der Herzentwicklung nur vorübergehend vorhanden, was die Ursache für die mangelnde Regenerationsfähigkeit des erwachsenen Organs ist. Die gezielte kardiale Differenzierung von hPSCs ist ein wirksames In-vitro-Modell der Kardiogenese. Die Vergänglichkeit der kardialen Vorläuferzellen erschwert jedoch auch die In-vitro-Untersuchung der Mechanismen, die ihre Entwicklung steuern. Es hat sich gezeigt, dass Histon-Acetyl-Transferasen (HATs) eine zentrale Rolle bei der Entwicklung und Homöostase des Herzens spielen. Trotz ihrer ubiquitären Rolle bei der Regulierung von Gennetzwerken fehlen systematische Studien zur Entwicklung und zu Krankheiten des Herzens. Wir sind speziell daran interessiert, die Wirkung von niedermolekularen Inhibitoren zu untersuchen, die in der Lage sind, HATs während der Herzentwicklung zu modulieren. Dies ist ein neuartiger Ansatz, um die Spezifikation und Differenzierung von hPSC-Kardiomyozyten zu manipulieren, insbesondere mit dem Ziel, stabilisierte kardiale Progenitoren (SCPs) zu etablieren, um die molekularen Eigenschaften dieser vorübergehend auftretenden und intensiv nachgefragten Zellen zu untersuchen.
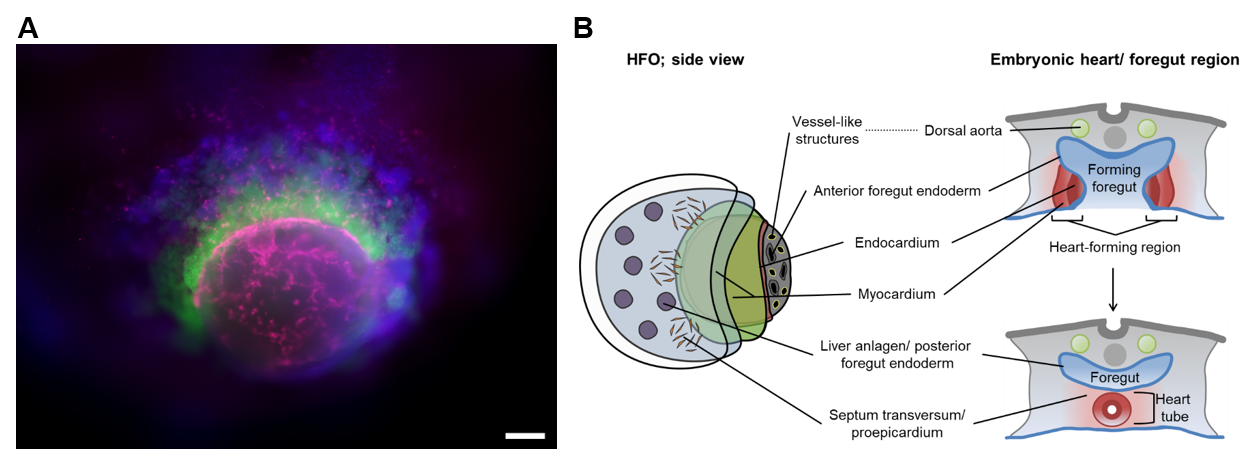
Herzbildende Organoide (HFOs): Als Reaktion auf geeignete Stimuli organisieren sich hPSCs selbst in 3D-Strukturen, die "Organoide" genannt werden und embryonalen Gewebemustern in vitro ähneln. Wir haben zum ersten Mal ein hochstrukturiertes Organoidmodell der frühen Herz-, Vorderdarm- und Gefäßentwicklung entwickelt, das als "Herzbildende Organoide" (HFOs) bezeichnet wird. HFOs bestehen aus Kardiomyozyten, Endokard-ähnlichen Zellen, Septum-Transversum-ähnlichen Anlagen, anteriorem und posteriorem Endodermgewebe des Vorderdarms und einem Gefäßnetzwerk (Abbildung 3). Neben der Entschlüsselung komplexer Mechanismen der menschlichen Organogenese in der Zellkulturschale kann das HFO-Modell auch als Ersatz für Tierversuche bei der Entdeckung und Entwicklung von Arzneimitteln verwendet werden, z. B. zur Bewertung der Teratogenität oder zum Screening von Arzneimitteln im Rahmen von Studien zu angeborenen Herzkrankheiten und zur Geweberegeneration.
Für die fortgeschrittene Analyse komplexer 3D-Strukturen über die Zeit/ in Echtzeit (4D) und die gezielte Manipulation und Verfolgung einzelner Zellen haben wir Projekte mit führenden Unternehmen im Bereich der Mikroskopie, z. B. LaVision BioTec/ Miltenyi Biotec (MoDiaNo), und mit akademischen Spezialisten für Femtosekundenlaser-Nanochirurgie- (FLN-) basierten Technologien (Kalies/ Heisterkamp, Leibniz Universität Hannover) eingerichtet.
Stabilisierte kardiale Progenitoren:
- Dorothee Bornhorst (Boston Children’s Hospital, Harvard University, USA)
- Salim Abdelilah-Seyfried (MHH/ Potsdam Universität)
- Lavanya Muthukrishnan Iyer (Universitätsmedizin Göttingen/ MDC Berlin)
- Laura C. Zelarayán-Behrend (Universitätsmedizin Göttingen)
- Dorota Zawada, Alessandra Moretti (Technische Universität München)
- Alexander Gödel (Karolinska-Institut, Schweden)
- Michael Morgan, Lange Lucas, Axel Schambach (MHH)
- Andreas Leffler (MHH)
- Gergana Dobreva (Anatomy and Developmental Biology Medical Faculty Mannheim, Heidelberg)
Herzformende Organoide:
- Simone Liebscher, Katja Schenke-Layland (Eberhard-Karls-Universität Tübingen)
- Jan Hegermann (MHH)
- Lena Nolte, Heiko Meyer (Laser Zentrum Hannover)
- Jeanne de la Roche, Stefan Thiemann, Christian Wahl-Schott (MHH)
- Stefan Kalies, Alexander Heisterkamp (Leibniz Universität Hannover)
- LaVision BioTec (Bielefeld)/ Miltenyi Biotec (Bergisch-Gladbach)
- Lothar Koch, Boris Chichkov (Leibniz Universität Hannover)
- Drakhlis L, Devadas SB, Zweigerdt R. Generation of heart-forming organoids from human pluripotent stem cells. Nat Protoc. 2021 Dec;16(12):5652-5672. doi: 10.1038/s41596-021-00629-8. Epub 2021 Nov 10. PMID: 34759383.
- Drakhlis L, Biswanath S, Farr CM, Lupanow V, Teske J, Ritzenhoff K, Franke A, Manstein F, Bolesani E, Kempf H, Liebscher S, Schenke-Layland K, Hegermann J, Nolte L, Meyer H, de la Roche J, Thiemann S, Wahl-Schott C, Martin U, Zweigerdt R. Human heart-forming organoids recapitulate early heart and foregut development. Nat Biotechnol. 2021 Feb 8. doi: 10.1038/s41587-021-00815-9. Epub ahead of print. PMID: 33558697.
- Müller D, Hagenah D, Biswanath S, Coffee M, Kampmann A, Zweigerdt R, Heisterkamp A, Kalies SMK. Femtosecond laser-based nanosurgery reveals the endogenous regeneration of single Z-discs including physiological consequences for cardiomyocytes. Sci Rep. 2019 Mar 6;9(1):3625. doi: 10.1038/s41598-019-40308-z.
- Gaspari E, Franke A, Robles-Diaz D, Zweigerdt R, Roeder I, Zerjatke T, Kempf H. Paracrine mechanisms in early differentiation of human pluripotent stem cells: Insights from a mathematical model. Stem Cell Res. 2018 Oct;32:1-7. doi: 10.1016/j.scr.2018.07.025. Epub 2018 Aug 18. PMID: 30145492.
- Koch L, Deiwick A, Franke A, Schwanke K, Haverich A, Zweigerdt R, Chichkov B.Laser bioprinting of human induced pluripotent stem cells-the effect of printing and biomaterials on cell survival, pluripotency, and differentiation. Biofabrication. 2018 Apr 25;10(3):035005. doi: 10.1088/1758-5090/aab981
- Kempf H, Olmer R, Haase A, Franke A, Bolesani E, Schwanke K, Robles-Diaz D, Coffee M, Göhring G, Dräger G, Pötz O, Joos T, Martinez-Hackert E, Haverich A, Buettner FFR, Martin U, Zweigerdt R. Bulk cell density and Wnt/TGFbeta signalling regulate mesendodermal patterning of human pluripotent stem cells. Nat Commun. 2016 Dec 9;7:13602. doi: 10.1038/ncomms13602. PMID: 27934856; PMCID: PMC5155150.
3. Modellierung von (angeborenen) Herzerkrankungen in der Zellkulturschale
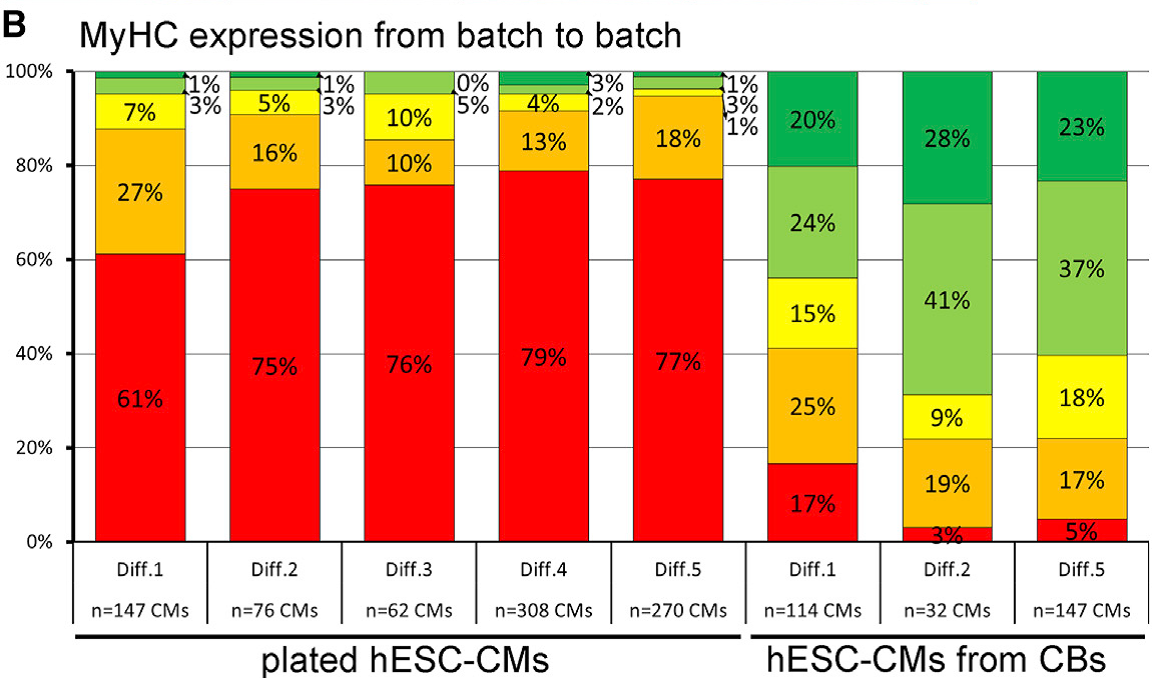
Aus hPSCs gewonnene Kardiomyozyten und kardiale Vorläuferzellen bieten ein großes Potenzial sowohl für neuartige Zelltherapien als auch für die In-vitro-Modellierung von Herzerkrankungen. Unter Anwendung unserer Technologien für die bioreaktorbasierte Massenproduktion von hPSC-Kardiomyozyten (die in jedem Screening-kompatiblen Kulturformat eingesetzt werden können) und der oben beschriebenen Erzeugung von anspruchsvolleren Herzbildenden Organoiden (HFOs) entwickeln wir Screening-Assays im kleinen bis mittleren Maßstab für die Modellierung von Herzerkrankungen in der Zellkulturschale. Zu unseren erfolgreichen Beispielen für diesen Ansatz gehören elektrophysiologische Störungen (z. B. Brugada-Syndrom aufgrund von Mutationen im SCN5A-Gen), die Inaktivierung kardialer Transkriptionsfaktoren (z. B. NKX2.5-Knock-out in HFOs) und jüngste Untersuchungen zur kardialen Hypertrophie in hPSC-abgeleiteten Kardiomyozyten auf der Basis pharmakologischer Stimuli oder genetischer Mutationen, z. B. in den MYH7- oder MYBPC3-Genloci (DFG-Projekt cMyBP-C-Mutationen FKZ: ZW 64/4-2). Hierfür spielt unter anderem die Reifung von hPSC-Kardiomyozyten ein zentrale Rolle, die die Expressionsmuster sarkomerer Proteine beeinflusst und unter anderem vom verwendeten Kulturformat abhängt (Figur 3).
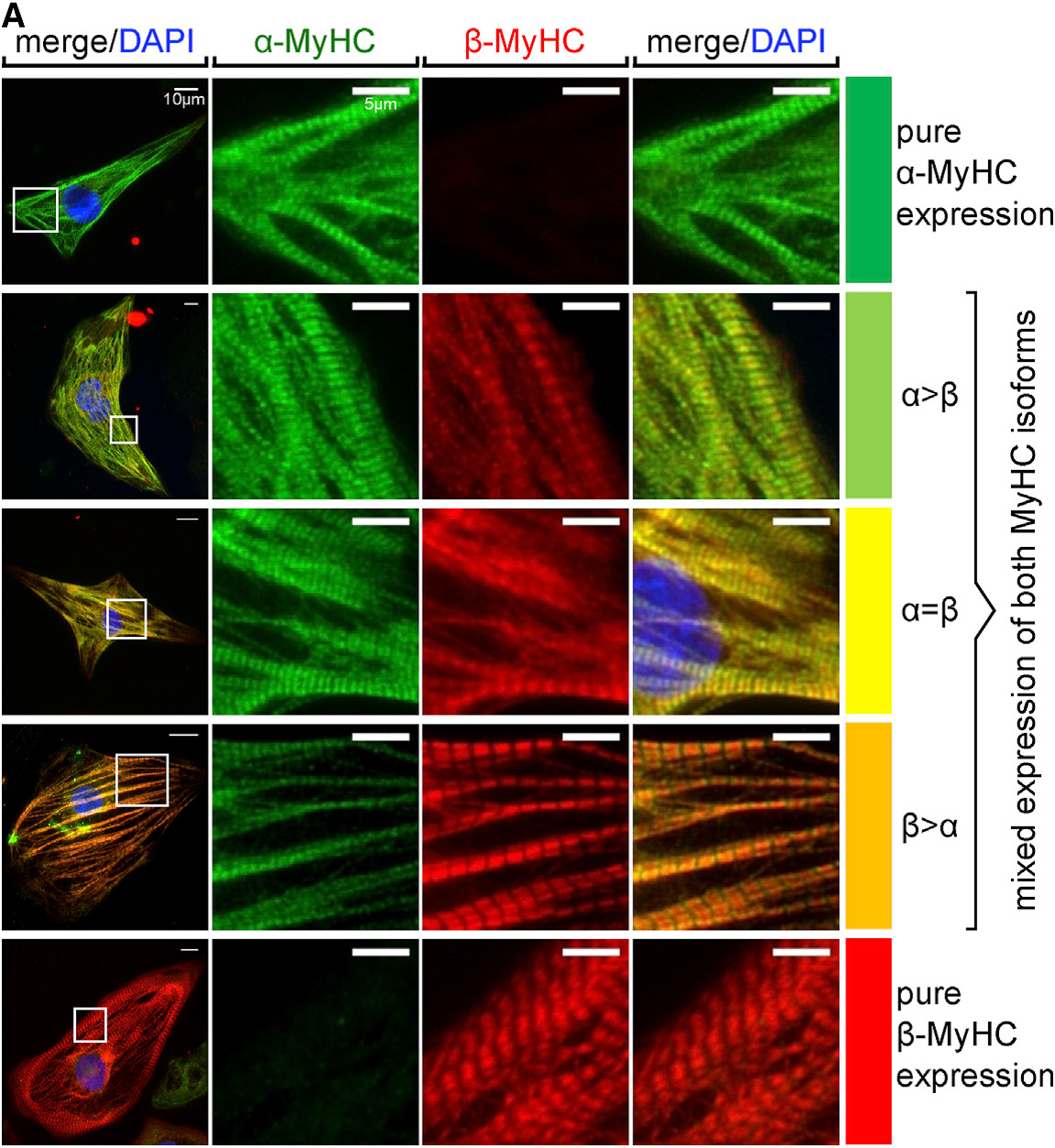
Für die Entwicklung eines Krankheitsmodells mit Vorhersagepotenzial ist es von größter Bedeutung, seine (biologischen) Grenzen zu verstehen. Dies gilt insbesondere, wenn man versucht, komplexe Pathologien wie die Herzhypertrophie zu modellieren. Länger andauernde mechanische Überlastung des Herzens oder Mutationen von Sarkomerproteinen können zu einem hypertrophen Umbau des Herzens führen. Um den zugrundeliegenden Mechanismus in vitro besser zu verstehen, entwickeln wir ein 3D-Modell der Herzhypertrophie unter Verwendung neurohormoneller Stimulation. Dieses Modell könnte für ein Mid-Throughput-Screening verwendet werden, um eine Reihe von Wirkstoffkandidaten zu testen, was wiederum den Einsatz von Tieren im Einklang mit den 3R-Prinzipien (Replacement, Reduction and Refinement) reduzieren könnte.
- Christian Bär (MHH )
- Martin Fischer (MHH)
- Bogdan Iorga (MHH)
- Theresia Kraft (MHH )
- Manuel H. Taft (MHH)
- Dietmar J. Manstein (MHH)
- Thomas Thum (MHH)
- Natalie Weber (MHH)
- Kai C. Wollert (MHH )
- Chatterjee S, Hofer T, Costa A, Lu D, Batkai S, Gupta SK, Bolesani E, Zweigerdt R, Megias D, Streckfuss-Bömeke K, Brandenberger C, Thum T, Bär C. Mol Ther. Telomerase therapy attenuates cardiotoxic effects of doxorubicin. 2021 Apr 7;29(4):1395-1410. doi: 10.1016/j.ymthe.2020.12.035. Epub 2021 Jan 1
- Merkert S, Wunderlich S, Beier J, Franke A, Schwanke K, Göhring G, Kraft T, Francino A, Zweigerdt R, Martin U. Generation of two iPSC clones (MHHi021-A and MHHi021-B) from a patient with hypertrophic cardiomyopathy with p.Arg723Gly mutation in the MYH7 gene. Stem Cell Res. 2021 Apr;52:102208. doi: 10.1016/j.scr.2021.102208. Epub 2021 Feb 3. PMID: 33578365.
- Latham SL, Weiß N, Schwanke K, Thiel C, Croucher DR, Zweigerdt R, Manstein DJ, Taft MH. Myosin-18B Regulates Higher-Order Organization of the Cardiac Sarcomere through Thin Filament Cross-Linking and Thick Filament Dynamics. Cell Rep. 2020 Sep 1;32(9):108090. doi: 10.1016/j.celrep.2020.108090.
- Hunkler HJ, Hoepfner J, Huang CK, Chatterjee S, Jara-Avaca M, Gruh I, Bolesani E, Zweigerdt R, Thum T, Bär C. The Long Non-coding RNA Cyrano Is Dispensable for Pluripotency of Murine and Human Pluripotent Stem Cells. Stem Cell Reports. 2020 Jul 14;15(1):13-21. doi: 10.1016/j.stemcr.2020.05.011. Epub 2020 Jun 11. PMID: 32531193
- Weber N, Kowalski K, Holler T, Radocaj A, Fischer M, Thiemann S, de la Roche J, Schwanke K, Piep B, Peschel N, Krumm U, Lingk A, Wendland M, Greten S, Schmitto JD, Ismail I, Warnecke G, Zywietz U, Chichkov B, Meißner J, Haverich A, Martin U, Brenner B, Zweigerdt R, Kraft T. Advanced Single-Cell Mapping Reveals that in hESC Cardiomyocytes Contraction Kinetics and Action Potential Are Independent of Myosin Isoform. Stem Cell Reports. 2020 May 12;14(5):788-802. doi: 10.1016/j.stemcr.2020.03.015. Epub 2020 Apr 16. PMID: 32302556; PMCID: PMC7220955.
- de la Roche J, Angsutararux P, Kempf H, Janan M, Bolesani E, Thiemann S, Wojciechowski D, Coffee M, Franke A, Schwanke K, Leffler A, Luanpitpong S, Issaragrisil S, Fischer M, Zweigerdt R. Comparing human iPSC-cardiomyocytes versus HEK293T cells unveils disease-causing effects of Brugada mutation A735V of NaV1.5 sodium channels. Sci Rep. 2019 Aug 1;9(1):11173. doi: 10.1038/s41598-019-47632-4. PMID: 31371804; PMCID: PMC6673693.
- Iorga B, Schwanke K, Weber N, Wendland M, Greten S, Piep B, Dos Remedios CG, Martin U, Zweigerdt R, Kraft T, Brenner B. Differences in Contractile Function of Myofibrils within Human Embryonic Stem Cell-Derived Cardiomyocytes vs. Adult Ventricular Myofibrils Are Related to Distinct Sarcomeric Protein Isoforms. Front Physiol. 2018 Jan 19;8:1111. doi: 10.3389/fphys.2017.01111. PMID: 29403388; PMCID: PMC5780405.
- Weber N, Schwanke K, Greten S, Wendland M, Iorga B, Fischer M, Geers-Knörr C, Hegermann J, Wrede C, Fiedler J, Kempf H, Franke A, Piep B, Pfanne A, Thum T, Martin U, Brenner B, Zweigerdt R, Kraft T. Stiff matrix induces switch to pure β-cardiac myosin heavy chain expression in human ESC-derived cardiomyocytes. Basic Res Cardiol. 2016 Nov;111(6):68. doi: 10.1007/s00395-016-0587-9. Epub 2016 Oct 14. PMID: 27743117.
- Viereck J, Kumarswamy R, Foinquinos A, Xiao K, Avramopoulos P, Kunz M, Dittrich M, Maetzig T, Zimmer K, Remke J, Just A, Fendrich J, Scherf K, Bolesani E, Schambach A, Weidemann F, Zweigerdt R, de Windt LJ, Engelhardt S, Dandekar T, Batkai S, Thum T. Long noncoding RNA Chast promotes cardiac remodeling. Sci Transl Med. 2016 Feb 17;8(326):326ra22. doi: 10.1126/scitranslmed.aaf1475.
